医師や研究者のための医学統計 2
下記をコピーして、先ほど開いたエディタの中に、ペーストして下さい
<作業ディレクトリの変更>
setwd("/Users/notake/Desktop/Rdata")
#notakeは私のコンピュータのことなので、
あなたのコンピュータ名を入力する必要があります
そのためには、コンソールの一番下にある
[履歴が次のファイルから読み込まれました /Users/xxx/ ]のxxxを打ち込みましょう
<エクセルファイルの読み込み>
mydata<-read.table("training1.txt", header=T)
#training1.txtというファイルを開くプログラム、1列目からデータが始まる場合には header=F、1列目に項目があるときはheader=Tとする
<ファイルをみる>
mydata
<データのサマリーをみる>
summary(mydata)
<カプラインマイヤーの計算>
library("survival")
mydata$time2<-Surv(mydata$OS,mydata$Censor)
(result<-survfit(time2~Treatment,data=mydata))
#ここでは、OS=期間、Censorは打ち切り(0:打ち切り、1:イベント)
<カプランマイヤーサマリーの提示>
summary(result)
<カプランマイヤーのグラフ>
par(lwd=2) #線の太さ
par(family="serif") #字体
plot(result,xlim=c(0,1825),xaxp=c(0,1825, 5), ylim=c(0,1), xlab="Time from Treatment (months)", ylab="Overall survival rate from relapse (proportion)",bty="l" ,tcl=0.2, lwd=2, lty=1,cex=0.5, cex.lab=1, cex.axis=1)
#col=は線の色、xlimはx軸の範囲(0,1825は0日-1825日まで)、xaxpは目盛りの両端と区切り、ylim=y軸の両端、main=はメインタイトル、cexは字の大きさ、xlab=x軸のラベル、bty=グラフの枠、tclは目盛りの向きと長さ、lwdは線の太さ、ltyは線の種類、cexは記号や字の大きさ
<Log rank test>
survdiff(Surv(OS,Censor)~Treatment,mydata)
#OS/Censor=期間/打ち切り、群分け:Treatmentで群分けして統計
<単変量Cox検定>
S.cox<-coxph( Surv(OS, Censor)~Treatment, data=mydata)
summary(S.cox)
<多変量解析Cox検定>
S.cox<-coxph( Surv(OS, Censor) ~Sex+over75+histology+Treatment,data=mydata)
summary(S.cox)
#mydataの中で、Sex、、、Treatmentについて検定
<t検定(ここではTreatment群間でのAgeの検定)>
t.test(mydata$Age~mydata$Treatment,var.equal=T) #student t検定
t.test(mydata$Age~mydata$Treatment) #Welchのt検定
bartlett.test(mydata$Age~mydata$Treatment) #bartlettの多群間検定
<カイ2乗検定 (数値を打ち込む場合)>
x<- matrix(c(35,224,14,119), ncol=2, byrow=T)
chisq.test(x) #カイ二乗検定
fisher.test(x) #フィッシャー検定
<MxN表の作り方 (通常はこちらでカイ二乗検定)>
x1<-table(mydata$Treatment,mydata$histology)
x2<- addmargins(x1)
x2
#Treatmentとhistologyの項目について多元表を作成
nc <- ncol(x2)
nr <- nrow(x2)
pcnt <- round(x2/x2[,nc]*100, 1)
pcnt
#%をみるなら
chisq.test(x1,correct=F)
#カイ二乗
fisher.test(x1)
#fisher検定
<logistic 回帰分析>
logisticmodel <- glm(Treatment ~ Sex+over75+stage+histology, family=binomial(logit), mydata)
logisticmodel
summary(logisticmodel)
exp(logisticmodel$coefficient)[-1]
exp(confint(logisticmodel))
エディタ上のプログラムをコンソールに呼び出す方法は、
「該当のプログラム」を選択してから、⌘+return(enter) です
はじめに、
Rdataフォルダのtraning1.txtファイルを読み取ります
①setwd("/Users/notake/Desktop/Rdata") を選択し ⌘+return
②mydata<-read.table("training1.txt", header=T) を選択し ⌘+return
ここで、読み取れませんなどのコメントが出る場合には、
exelに打ち込んだデータにスペースがないか、ファイル名の大文字/小文字
ファイルの保存場所、ファイルの保存形式(タブ付きテキスト)を確認しましょう
つぎに、exelデータが読み取れているかを確認します
③mydata
④summary(mydata) どちらか(両方でも良い)を選択し ⌘+return
ここまでしていて、データが上手く表示されない場合
原因が分かりませんが、このホームページのプログラムをエディタにペーストしても上手く使用できないことがあります(プログラムなどに疎いので、ここら辺の理由は分かりません。)
その場合は、同じ文章をエディタに書き込み、選択し ⌘+return して下さい
特に
mydata<-read.table("training1.txt", header=T) を書き直すと良いようです
(以後、やはり読み取りにくいプログラムがありますので、上手く表示されない時はプログラムを手打ちしてみて下さい。手打ちしたエディタを保存しておけば、その後は大丈夫です)

logrank検定します
survdiff(Surv(OS,Censor)~Treatment,mydata)
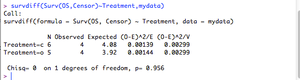
ここでは、p=0.956と有意差はありません
